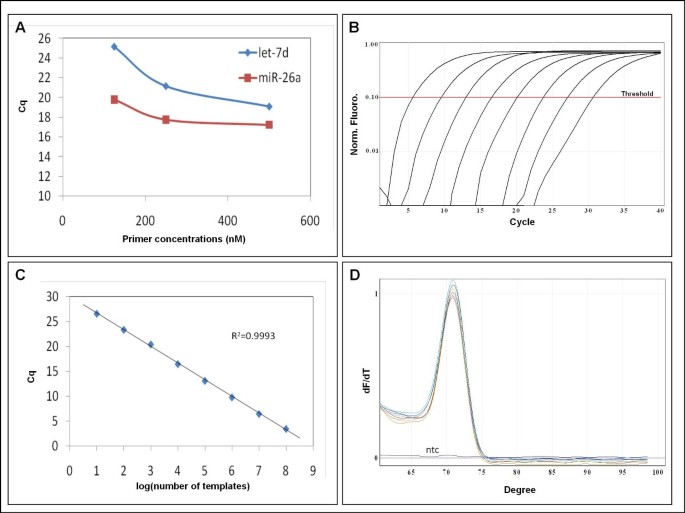
La PCR cuantitativa de muestras biológicas se realizó en 10 μl de volumen total con 1 μl de ADNc diluido 8-10 veces, 5 μl de 2x mezcla maestra de PCR QuantiFast SYBR Green (Qiagen, Alemania), 250 nM de cada cebador o 2 μl microRNA LNATM cebador conjuntos (Exiqon, Dinamarca). Se realizaron curvas estándar con diluciones de 10 veces para todos los ensayos para calcular la eficiencia de qPCR.(1)
Se usaron las mismas condiciones de PCR para las plantillas sintéticas, excepto que se usó 1 μl de plantilla sintética en 2 ng / μl de ADN de esperma de salmón (Sigma, EE. UU.) En TE en lugar de ADNc. Se utilizó 2x Brilliant III Ultra-Fast QPCR Master Mix (Agilent, EE. UU.)
Las condiciones de ciclismo fueron 95 ° C durante 5-10 min seguidas de 40 ciclos de 95 ° C durante 10-30 segundos y 60 ° C 30-60 segundos. Se realizó un análisis de la curva de fusión (60 ° C a 99 ° C) después del perfil térmico para garantizar la especificidad en la amplificación.
El QPCR de muestras biológicas se realizó en una máquina MX3000P (Stratagene, EE. UU.) Y las reacciones que contienen plantillas sintéticas se realizaron en un Rotorcycler (Qiagen, Alemania). Los cebadores enriquecidos con LNA fueron conjuntos de cebadores de microRNA LNA ™ PCR diseñados por Exiqon (Dinamarca)(1)
La cuantificación se basó en la determinación del ciclo de cuantificación (Cq) y la eficiencia de la PCR se calculó a partir de la porción logarítmica lineal de las curvas estándar(1)
Referencias Bibliográficas:
- Balcells, I., Cirera, S. & Busk, P.K. Specific and sensitive quantitative RT-PCR of miRNAs with DNA primers. BMC Biotechnol 11, 70 (2011) doi:10.1186/1472-6750-11-70